近期,华大智造针对DNBSEQ平台的甲基化建库与测序进行了全面升级,推出新一代建库产品MGIEasy 全基因组甲基化文库制备试剂盒V3.0(MGIEasy Whole Genome Methylation Sequencing Library Prep Kit V3.0,以下简称WGMS V3.0)。WGMS V3.0以双链加接头方法为基础,对经物理打断方法获得的DNA片段进行建库,并适配酶法转化,将非甲基化的胞嘧啶(C)转化为尿嘧啶(U),搭配DNBSEQ平台,能够实现单碱基精度地解析DNA甲基化状态,构建精细的全基因组甲基化图谱。WGMS V3.0可为大人群表观组研究、表观遗传基础科研、动植物研究、疾病甲基化标志物筛选、药物研发等多个领域提供高效而准确的全基因组甲基化建库方法。
当前,WGMS V3.0已适配MGISEQ-2000平台,结合MGISEQ-2000平台的升级算法,测序时可以不添加平衡文库,实现0平衡文库的纯甲基化文库测序。此外,华大智造研发团队针对WGMS V3.0适配DNBSEQ-T7已启动最后的稳定性测试,更多实测数据将持续分享,敬请期待。
产品亮点
- 可获得更大文库插入片段,支持PE150测序
- 起始量范围广,可兼容100~1000ng gDNA建库起始量,5~20ng cfDNA建库起始量
- 兼容多种样本类型,包括血液、新鲜组织、细胞样本、FFPE样本、血浆样本,以及常见动植物gDNA样本
- 建库采用磁珠纯化方法,全流程适配自动化设备
- 搭配DNBSEQ平台测序数据利用率高,基因组覆盖度高,甲基化检测一致性高
实测数据
1、0平衡文库,数据利用率高,轻松实现“1库1 lane”
采用3个不同批次的WGMS V3.0构建NA12878标准品的DNA甲基化文库,不加平衡文库,平行上机MGISEQ-2000 ECR7.0(搭配SM测序试剂)和ECR7.1(搭配SM 2.0测序试剂),PE150测序,单lane reads产出基本稳定在400 M左右,单个文库可获得约120G的原始下机数据,可轻松实现“1库1 lane”,且测序质量良好,SM测序试剂Q30稳定在89%左右,SM2.0测序试剂Q30稳定在94%左右,Q40稳定在87%左右(表1),Read1和Read2的测序碱基分布线几乎稳定在一条水平线上,测序质量高(图1)。
表1. WGMS V3.0适配MGISEQ-2000平台测序数据
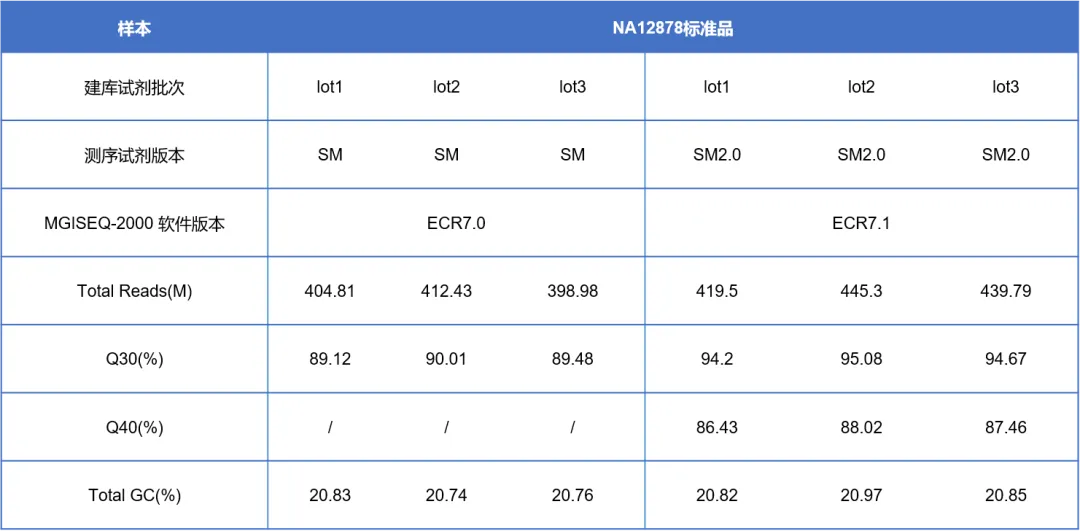
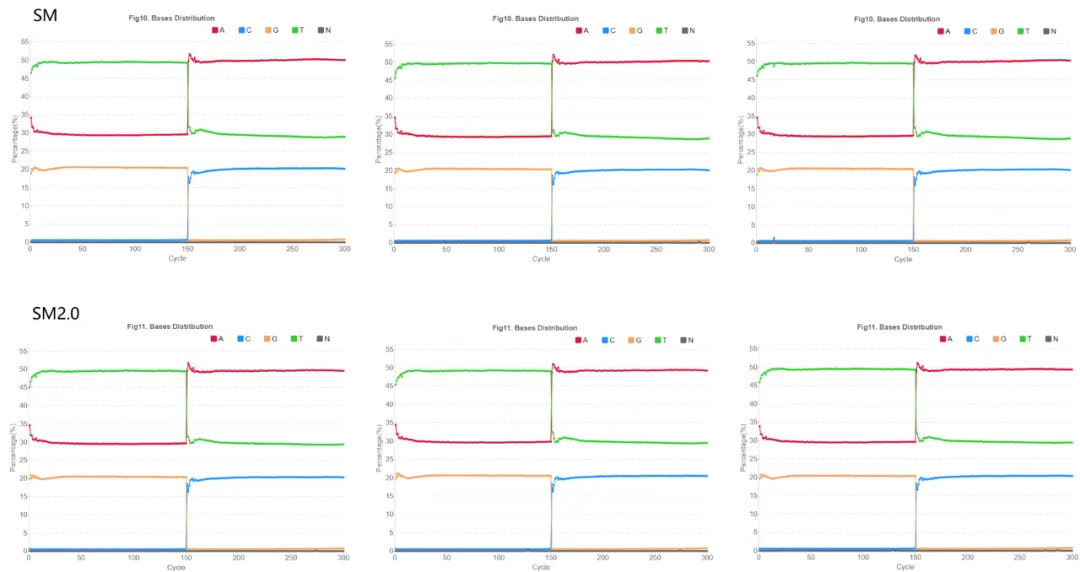
图1. Read1和Read2的测序碱基分布
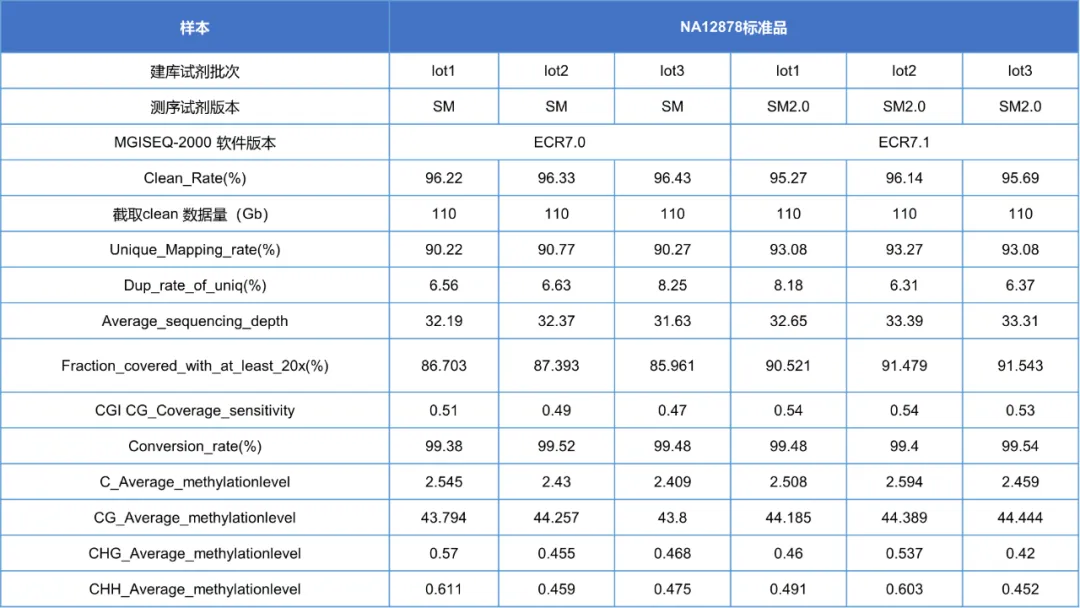
不同文库的MGISEQ-2000 PE150测序数据clean rate≥95%,截取110Gb clean data进行分析,平均测序深度均≥30x,搭配ECR7.0软件版本测序仪的全基因组20×coverage≥85%,ECR7.1软件版本测序仪的全基因组20×coverage≥90%。质控文库Lambda DNA非甲基化C转U的转化率≥99%,不同批次建库试剂盒及不同测序试剂版本下的NA12878标准品DNA的全基因组甲基化率保持在相当的水平(43~45%)。
表2. WGMS V3.0适配MGISEQ-2000平台的测序数据
3、文库插入片段大
试剂盒适配转化损伤小的酶法转化,增大了文库插入片段长度,更好地支持PE150测序。不同批次建库试剂盒构建的NA12878标准品DNA甲基化文库的插入片段(主峰约280bp)表现一致。
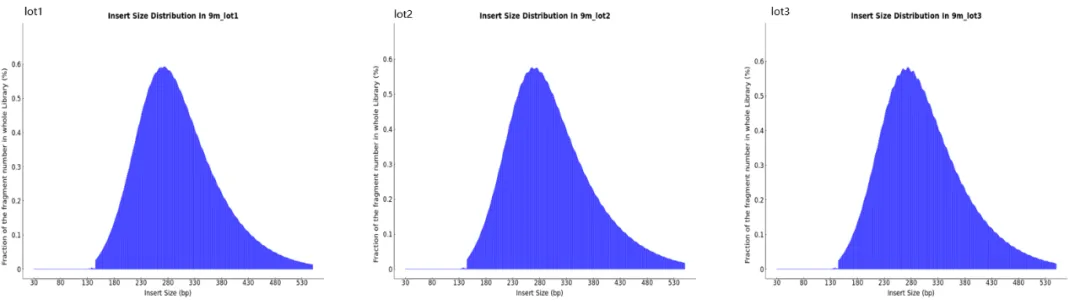
图2. DNA甲基化文库的插入片段大小
4、M-bias低
试剂盒优化了末端修复反应体系和步骤,降低了Reads末端M-bias碱基个数。SM2.0版测序试剂搭配ECR7.1软件版本所获结果显示,不同批次建库试剂盒构建的NA12878标准品DNA甲基化文库的M-bias表现一致,Bottom Strand 和Top Strand的Read1和Read2甲基化率几乎重合。
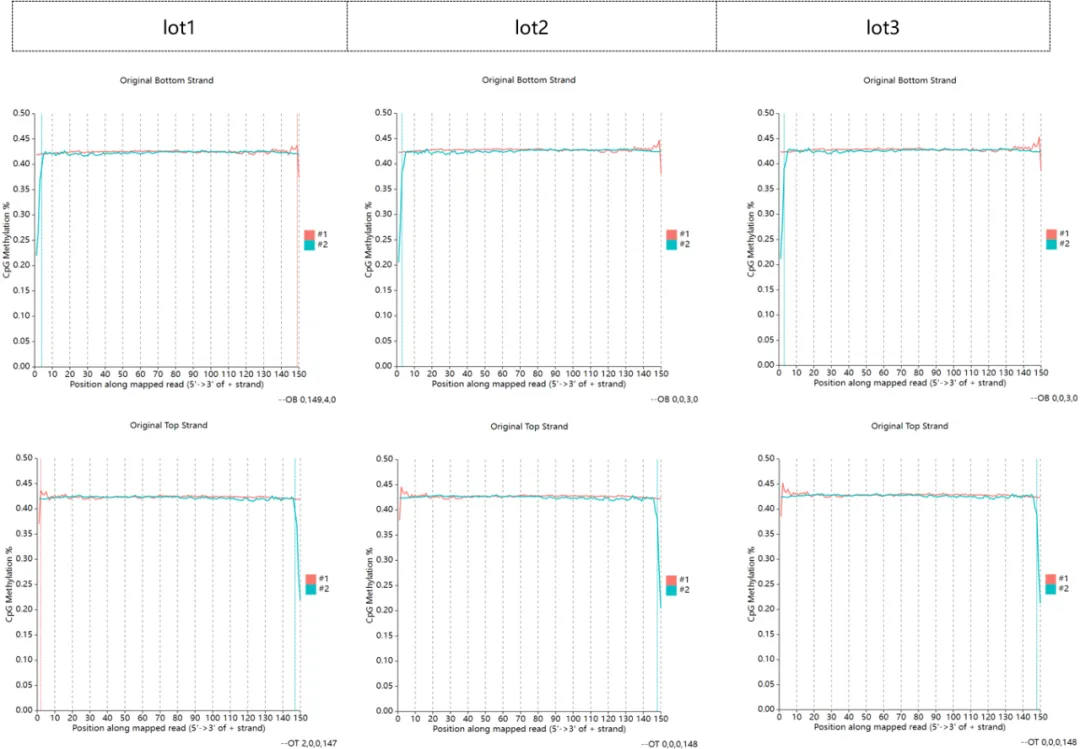
图3. M-bias图
5、甲基化检测一致性高
分析各文库在4×测序深度下检测到的独有和共有的CpG位点,SM2.0版测序试剂搭配ECR7.1软件版本所获结果显示,3个批次试剂盒所建的甲基化文库的CpG位点覆盖一致性>95%(图4),不同建库试剂盒批次间的甲基化率一致性可高达94%(图5)。
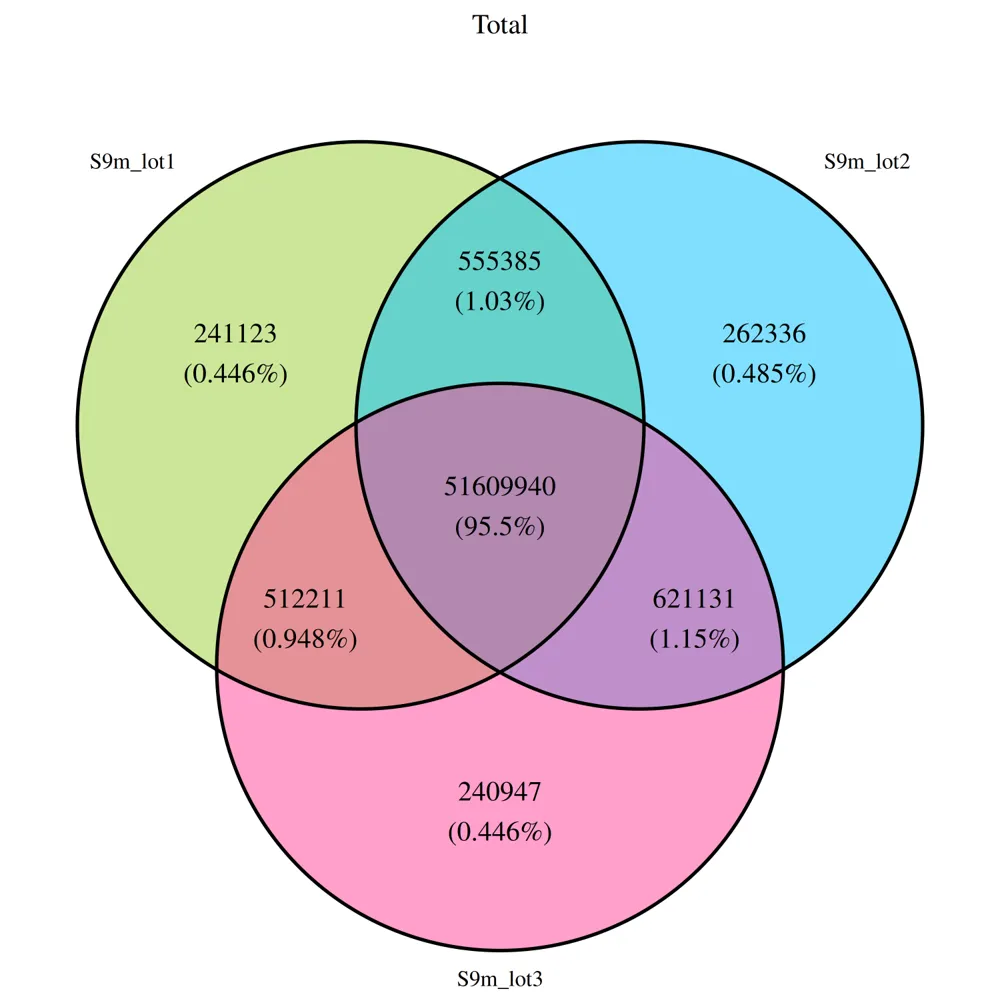
图4. lot1、lot2、 lot3 文库的CpG位点覆盖一致性
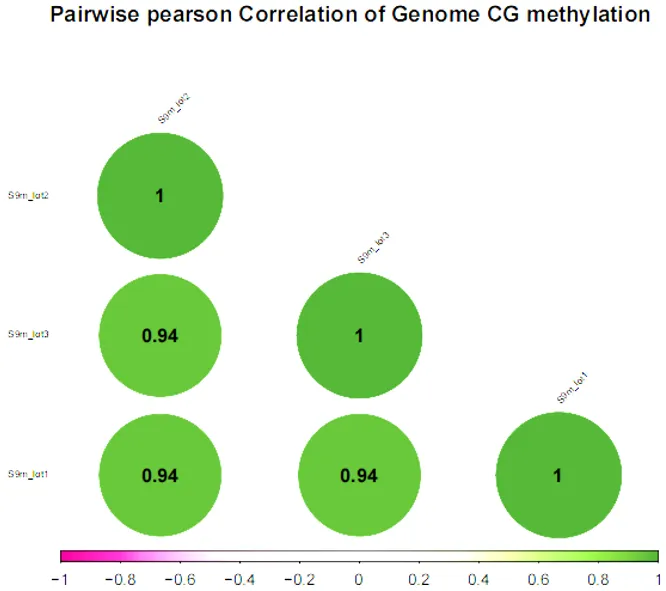
图5. lot1、lot2、 lot3 文库间的CpG位点检测相关性分析
产品订购信息




 基因测序仪业务 SEQ ALL
基因测序仪业务 SEQ ALL











 核心技术
核心技术 热门应用
热门应用 组学研究
组学研究 文档下载
文档下载 已发表文章
已发表文章 用户成功案例
用户成功案例 在线数据集
在线数据集 多功能中心
多功能中心 客户中心
客户中心 在线支持
在线支持 关于智造
关于智造 专题推荐
专题推荐